Bioimaging-Methoden ermöglichen einen tieferen Einblick in die molekularen und zellulären Prozesse des menschlichen Körpers als je zuvor. Das Verfahren des Identifizierens und Nachweisens der Grenzen bestimmter Interessensbereiche wie Einzelzellen in der mikroskopischen Bildgebung wird Segmentierung genannt. In den letzten Jahren konnte die Bildsegmentierung mit Deep Learning in der biomedizinischen Forschung große Erfolge feiern. Der Algorithmus lernt in der Regel durch eine große Menge „ground truth“, Trainingsdaten, die von Menschen annotiert wurden. Für dieses überwachte Lernverfahren müssen Forschende eine große Datenmenge markieren. Obwohl nicht überwachte Verfahren zum Trainieren von Deep-Learning-Modellen ohne menschliche Annotation existieren, ist deren Leistung oft ungenauer als die von überwachten Verfahren und somit nicht ausreichend für die biomedizinische Forschung. Darum ist es das Ziel der Forschenden, die am Projekt »Smart Human-in-the-loop Segmentation« arbeiten, ein leistungsstarkes Deep-Learning-Modell zu entwickeln, das mit minimaler menschlicher Arbeit auskommt.
Ein Workflow mit Open Source Deep Learning ermöglicht eine effizientere Bildsegmentierung
Das Gesamtverfahren generalisiert den iterativen Deep-Learning-Workflow des Allen Cell and Structure Segmenter und entwickelt ihn weiter. Der erwähnte Segmentierer ist ein Toolkit für die 3D-Segmentierung von intrazellulären Strukturen in Fluoreszenzmikroskopiebildern, das von Dr. Jianxu Chen und dem Allen Institute for Cell Science entwickelt wurde. Der erste Schritt der »Smart Human-in-the-loop Segmentation« ist die Entwicklung von effektiven Strategien zum automatischen Auswählen repräsentativer Proben für die anfängliche Kuration oder Annotation basierend auf der bisherigen Arbeit des Teams an suggestiver Annotation, einem Deep-Active-Learning-Framework, das ein vollständig konvolutionales Netzwerk mit aktivem Lernen kombiniert. Als Nächstes werden einige Proben für eine grobe manuelle Annotation durch die Forschenden ausgewählt. Diese werden für das anfängliche Training des Algorithmus für die Segmentierung von Bildern, der anschließend auf alle Bilder in dem Datensatz angewandt wird, verwendet. Danach werden einige Bilder manuell geprüft und zum Trainieren des Algorithmus in den Workflow eingebracht.
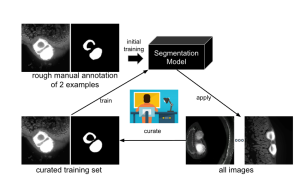
Übersicht des Human-in-the- Loop- Segmentierungsworkflows für die automatisierte Analyse des Volumens von B-Zell- Follikeln und der T-Zell-Zone in einem Peyer-Plaque, einer Gruppe aus Lymphfollikeln in der Schleimhautmembran des Dünndarms. Die Bilder auf der linken Seite sind die Rohbilder der Lichtblattfluoreszenzmikrosko pie und die auf der rechten Seite sind die Bilder nach der Segmentierung.
© ISAS / Jianxu Chen
Für jede Iteration entwickeln die Forschenden Verfahren, um es dem Deep-Learning-Modell zu ermöglichen, potenzielle Fehler automatisch zu identifizieren und eine menschliche Kuration einzuleiten. Schließlich werden effektive aktive Lernmethoden und kontinuierliche Lernmethoden entwickelt, um das sogenannte katastrophale Vergessen zu verhindern. Dies ist der drastische Verlust von zuvor erlernten Informationen nach dem Erlernen einer neuen Aufgabe im iterativen Deep-Learning-Workflow. Dies ermöglicht eine genaue Bildsegmentierung ohne viel menschliches Eingreifen. Langfristig soll der entwickelte Algorithmus als Open-Source-Software mit grafischem Nutzeroberflächentool für die breitere biomedizinische Bildanalyse bereitgestellt werden.
Teilen
Ausgewählte Publikationen
Npj imaging, Bd. 2025, Nr. 3, 2025
Pan Z, Sonneck J, Nagel D, Hasenberg A, Gunzer M, Shi Y, Chen J.
AutoQC-Bench: a diffusion model and benchmark for automatic quality control in high-throughput microscopy
https://doi.org/10.1038/s44303-025-00117-8
Nature Computational Science, Bd. 5, Nr. 11, 2025, S. 1041-1050
Dai G, Zhang R, Wuwu Q, Tseng C, Zhou Y, Wang S, Qian S, Lu M, Tuz AA, Gunzer M, Huang T, Chen J, Zhang S.
Implicit neural image field for biological microscopy image compression
https://doi.org/10.1038/s43588-025-00889-4
IEEE Transactions on Medical Imaging, 2025, S. 1-1
Liang P, Ding Y, Zhang Y, Chen J, Zheng H, Wang H, Zhang Y, Meng G, Weninger T, Niemier M, Sharon Hu X, Chen DZ.
Cell Instance Segmentation: The Devil Is in the Boundaries
https://doi.org/10.1109/TMI.2025.3621093
Npj imaging, Bd. 3, Nr. 1, 2025, S. 29
Cao J, Wenzel J, Zhang S, Lampe J, Wang H, Yao J, Zhang Z, Zhao S, Zhou Y, Chen C, Schwaninger M, Yang J, Chen DZ, Chen J.
Rethinking deep learning in bioimaging through a data centric lens
https://doi.org/10.1038/s44303-025-00092-0
Journal of Neuroinflammation, Bd. 22, Nr. 1, 2025
Christ R, Siemes D, Zhao S, Widera L, Spangenberg P, Lill J, Thiebes S, Bottek J, Borgards L, Pinho AG, Silva NA, Monteiro S, Jorch SK, Gunzer M,…
Inhibition of tumour necrosis factor alpha by Etanercept attenuates Shiga toxin-induced brain pathology
https://doi.org/10.1186/s12974-025-03356-z
Biomedizinische Technik / Biomedical engineering, Bd. 70, Nr. 1, 2024, S. 71-90
Li J, Zhou Z, Yang J, Pepe A, Gsaxner C, Luijten G, Qu C, Zhang T, Chen X, Li W, Wodzinski M, Friedrich P, Xie K, Jin Y, Ambigapathy N, Nasca E,…
MedShapeNet - a large-scale dataset of 3D medical shapes for computer vision.
https://doi.org/10.1515/bmt-2024-0396
Computational Mechanics, Bd. 76, Nr. 1, 2025, S. 117-146
Shi R, Yang H, Chen J, Hackl K, Avril S, He Y.
Deep learning without stress data on the discovery of multi-regional hyperelastic properties
https://doi.org/10.1007/s00466-024-02591-0
Biological Imaging, Bd. 4, 2024, S. e16
Zhou Y, Sollmann J, Chen J.
Deep-learning-based image compression for microscopy images: An empirical study
https://doi.org/10.1017/S2633903X24000151
JMIR Serious Games, Bd. 12, 2024, S. e52785
Egger J, Gsaxner C, Luijten G, Chen J, Chen X, Bian J, Kleesiek J, Puladi B.
Is the Apple Vision Pro the Ultimate Display? A First Perspective and Survey on Entering the Wonderland of Precision Medicine
https://doi.org/10.2196/52785
Nature Methods, Bd. 21, Nr. 8, 2024, S. 1390-1393
Zhang S, Dai G, Huang T, Chen J.
Multimodal large language models for bioimage analysis
https://doi.org/10.1038/s41592-024-02334-2
Journal of Cerebral Blood Flow and Metabolism, Bd. 45, Nr. 1, 2024, S. 171-186
Hagemann N, Qi Y, Mohamud Yusuf A, Li A, Zhang X, Spangenberg P, Squire A, Doeppner TR, Jin F, Zhao S, Chen J, Mosig A, Gunzer M, Hermann DM.
Arterial specification precedes microvascular restitution in the peri-infarct cortex that is driven by small microvessels
https://doi.org/10.1177/0271678X241270407
Dagstuhl Reports, Bd. 14, Nr. 1, 2024, S. 90-107
Chen J, Jug F, Rafelski S, Zhang S.
The Emerging Issues in Bioimaging AI Publications and Research (Dagstuhl Seminar 24042)
https://doi.org/10.4230/DagRep.14.1.90
Journal of Imaging Informatics in Medicine, Bd. 38, Nr. 1, 2024, S. 646-655
Krieger K, Egger J, Kleesiek J, Gunzer M, Chen J.
Multisensory Extended Reality Applications Offer Benefits for Volumetric Biomedical Image Analysis in Research and Medicine
https://doi.org/10.1007/s10278-024-01094-x
2024 IEEE International Symposium on Biomedical Imaging (ISBI), 2024
Zhou Y, Zhao S, Sonneck J, Chen J.
2D Label-Free Prediction of Multiple Organelles Across Different Transmitted-Light Microscopy Images with Bag-of-Experts
https://doi.org/10.1109/ISBI56570.2024.10635298
2024 IEEE International Symposium on Biomedical Imaging (ISBI), 2024
Yao J, Hagemann N, Xiong Q, Chen J, Hermann DM, Chen C.
Topological Analysis of Mouse Brain Vasculature via 3d Light-Sheet Microscopy Images
https://doi.org/10.1109/ISBI56570.2024.10635226
Nature Cardiovascular Research, Bd. 3, Nr. 5, 2024, S. 525-540
Tuz AA, Ghosh S, Karsch L, Ttoouli D, Sata SP, Ulusoy ?, Kraus A, Hoerenbaum N, Wolf J, Lohmann S, Zwirnlein F, Kaygusuz V, Lakovic V, Tummes H, Beer…
Stroke and myocardial infarction induce neutrophil extracellular trap release disrupting lymphoid organ structure and immunoglobulin secretion
https://doi.org/10.1038/s44161-024-00462-8
Nature Methods, Bd. 21, Nr. 2, 2024, S. 182-194
Reinke A, Tizabi MD, Baumgartner M, Eisenmann M, Heckmann-Nötzel D, Kavur AE, Rädsch T, Sudre CH, Acion L, Antonelli M, Arbel T, Bakas S, Benis A,…
Understanding metric-related pitfalls in image analysis validation
https://doi.org/10.1038/s41592-023-02150-0
GigaScience, 2024
Sonneck J, Zhou Y, Chen J.
MMV_Im2Im: an open-source microscopy machine vision toolbox for image-to-image transformation
https://doi.org/10.1093/gigascience/giad120
Nature Methods, Bd. 21, Nr. 3, 2024, S. 368-369
Zhou Y, Cao J, Sonneck J, Banerjee S, Dörr S, Grüneboom A, Lorenz K, Zhang S, Chen J.
EfficientBioAI: making bioimaging AI models efficient in energy and latency
https://doi.org/10.1038/s41592-024-02167-z
Nature Communications, Bd. 14, 2023, S. 1-13
Cibir Z, Hassel J, Sonneck J, Kowitz L, Beer A, Kraus A, Hallekamp G, Rosenkranz M, Raffelberg P, Olfen S, Smilowski K, Burkard R, Helfrich I, Tuz…
ComplexEye:a multi-lens array microscope for high-throughput embedded immune cell migration analysis
https://doi.org/10.1038/s41467-023-43765-3
Nature Machine Intelligence, Bd. 5, 2023, S. 687-698
Jia Z, Chen J, Xu X, Kheir J, Hu J, Xiao H, Peng S, Hu X, Chen D, Shi Y.
The importance of resource awareness in artificial intelligence for healthcare
https://doi.org/10.1038/s42256-023-00670-0
Nature Methods, Bd. 20, 2023, S. 968-970
Chen J, Viana M, Rafelski S.
When seeing is not believing: application-appropriate validation matters for quantitative bioimage analysis
https://doi.org/10.1038/s41592-023-01881-4
Cell Reports Methods, Bd. 3, Nr. 3, 2023, S. 100436
Spangenberg P, Hagemann N, Squire A, Förster N, Krauß SD, Qi Y, Mohamud Yusuf A, Wang J, Grüneboom A, Kowitz L, Korste S, Totzeck M, Cibir Z, Tuz AA,…
Rapid and fully automated blood vasculature analysis in 3D light-sheet image volumes of different organs
https://doi.org/10.1016/j.crmeth.2023.100436
IEEE Transactions on Medical Imaging, Bd. 41, Nr. 10, 2022, S. 2582-2597
Liang P, Zhang Y, Ding Y, Chen J, Madukoma CS, Weninger T, Shrout JD, Chen DZ.
H-EMD: A Hierarchical Earth Mover's Distance Method for Instance Segmentation
https://doi.org/10.1109/TMI.2022.3169449
Immunological Reviews, Bd. 306, Nr. 1, 2021, S. 293-303
Grüneboom A, Aust O, Cibir Z, Weber F, Hermann DM, Gunzer M.
Imaging innate immunity
https://doi.org/10.1111/imr.13048
