Das Bestimmen der Bewegung, Ausrichtung und des Wachstums von Zellen spielt eine entscheidende Rolle für das Verständnis der Mechanismen von Gesundheit und Erkrankung. Darum ist die Fähigkeit, Zellen im Zeitraffer von zwei- oder dreidimensionalen Mikroskopiebildern zu verfolgen ein wichtiger Teil der biomedizinischen Forschung: Dadurch können wichtige Einblicke in die Entwicklung von beispielsweise verschiedenen Krebsarten bereitgestellt werden. Im vergangenen Jahrzehnt wurden verschiedene Algorithmen und Tools für das Zell-Tracking entwickelt. Forschende in der Biomedizin haben jedoch nach wie vor mit dem Extrahieren von relevanten Informationen aus ihren Zeitraffer-Mikroskopiedaten zu kämpfen. Der Hauptgrund dafür ist, dass es keine universelle Lösung für die Verarbeitung verschiedener Zeitrafferarten mit beispielsweise verschiedenen Bildgebungseinstellungen, Zelltypen, Bewegungsarten oder Analyseanforderungen gibt. Darum will das Forschungsprojekt »Zell-Tracking in Mikroskopieaufnahmen« (Cell Tracking in Microscopy Images) neue, anspruchsvolle Algorithmen für den Zellnachweis und das Zell-Tracking entwickeln, um einige der schwierigsten Tracking-Probleme zu adressieren.
Tracking-Probleme, bei denen bisherige Verfahren daran scheitern, zufriedenstellende Ergebnisse zu liefern, sollen in diesem Projekt von Forschenden gelöst werden. Laut ihnen kann der Tracking-Prozess unter anderem durch ein Überlappen oder Verklumpen der Zellen besonders erschwert werden. Darüber hinaus kann sich die Bewegung der Zellen in verschiedenen Umgebungen, zum Beispiel unter Bedingungen mit verschiedenen Wirkstoffen, signifikant unterscheiden. Aufgrund dessen muss der Tracking-Algorithmus allgemein genug sein, um verschiedene mögliche Bewegungsmuster zu verfolgen. Die Forschenden nehmen sich dieser Herausforderungen durch modernste Deep-Learning-Verfahren an.
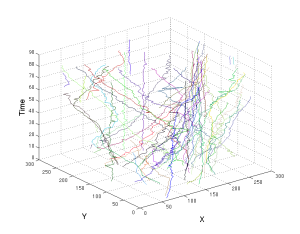
Eine visuelle Darstellung der Bewegungsbahn von Zellen im Raum mit der Zeit. Jede farbige Bewegungsbahn gehört zu einer anderen Zelle. Die x- und y-Achsen kennzeichnen die räumliche Position der Zellen in einem Bild.
© ISAS / Jianxu Chen
Open-Source-Zugriff auf Human-in-the-loop-Zell-Tracking mittels napari-Plug-in
Um die verbesserte Analyse der Mikroskopiebilder für Forschende weltweit frei verfügbar zu machen, entwickelt das Projektteam ein Human-in-the-loop-Zell-Tracking-Plug-in für die Bildanalyseplattform napari. Herkömmliche Tools legen den Schwerpunkt meist auf die Zellsegmentierung- und Tracking-Algorithmen, die vollautomatisiert sind. In vielen Situationen sind die Ergebnisse der vollautomatisierten Algorithmen jedoch nicht zufriedenstellend. Daher ist es das Ziel der Forschenden, Menschen durch eine praktische grafische Benutzeroberfläche wieder in den Datenanalyseprozess einzubeziehen. Das Ziel ist die Entwicklung eines automatisierten Zell-Tracking-Algorithmus, der die bestmöglichen Ergebnisse liefern kann, selbst bei herausfordernden Aufgaben. Gleichzeitig soll ihre Zell-Tracking-Lösung es Forschenden in der Biomedizin ermöglichen, in die automatisierte Analyse einzugreifen, potenzielle Fehler zu korrigieren und die Ergebnisse gemäß der gewünschten Genauigkeit zusammenzustellen. Die „Human-in-the-Loop Cell Tracking“-Software wird aus drei Modulen (Segmentierung, Tracking und Analyse) bestehen. Durch den Einsatz von künstlicher Intelligenz besteht die Aufgabe des Plug-ins darin, die Ergebnisse der napari-Bildanalyse zu optimieren und Machine Learning zu nutzen, um den Algorithmus mit von Menschen bereitgestellten Daten zu trainieren.