Dortmund, 20. Dezember 2023
Vor Jahrhunderten öffnete die Erfindung des Mikroskops ein Fenster zu einer winzigen, bis dato unsichtbaren Welt. Seitdem hat sich nicht nur die Qualität der Bilder, sondern auch die Art und Weise, wie wir sie interpretieren, stark verändert. Statt nur optisch in die Tiefen kleinster Strukturen einzudringen, können wir biologische Prozesse heute mithilfe moderner Bildanalyseverfahren in präzise Zahlen übersetzen. Doch damit diese Übersetzung gelingt, müssen sich alle Forschenden zunächst dieselben Fragen stellen: Was wollen wir messen? Wie genau müssen diese Messungen sein, damit wir sie sinnvoll interpretieren können?
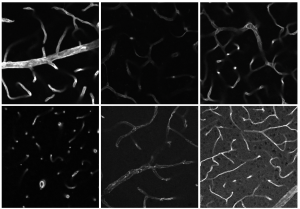
Moderne Mikroskope erzeugen enorme Datenmengen. Ein Lichtblattfluoreszenz-Mikroskop beispielsweise, produziert am ISAS pro Probe durchschnittlich weit über 500 hochauflösende Bilder. Um diese Datenvielfalt zu bewältigen und im Anschluss effizient auszuwerten, braucht es innovative Ansätze in der Datenanalyse. Hierbei kommen spezielle Softwares oder Bildanalyseverfahren zum Einsatz, zunehmend auch mithilfe Künstlicher Intelligenz (KI). Einen universell anwendbaren Ansatz für die Bildanalyse gibt es jedoch nicht. „Genauso wie es kein 'one size fits all' Mikroskop gibt, erfordern verschiedene Forschungsvorhaben maßgeschneiderte und auf die spezifischen Anforderungen abgestimmte Bildanalyse-Workflows oder Softwares“, erläutert Dr. Jianxu Chen. Er leitet die Nachwuchsgruppe AMBIOM – Analysis of Microscopic BIOMedical Images, die am ISAS an genau solchen KI-basierten biomedizinischen Bildanalysealgorithmen forscht.

Bei der Checkliste geht es um das Gleichgewicht zwischen Analyse und Genauigkeit. Diese Abbildung hat Dr. Jianxu Chen am 13. Dezember 2023 mittels ChatGPT, Modell „DALL-E“ erstellt. Details hierzu inkl. Prompt siehe Seitenende.
© ChatGPT
Eine Checkliste, um Bioimage-Analyseergebnisse zu validieren
Die Herausforderung bei der Bildanalyse besteht darin, sicherzustellen, dass die gewonnenen Erkenntnisse am Ende nicht nur quantitativ oder statistisch genau, sondern auch biologisch relevant für die Fragestellung sind. „Dafür sollten Forschende die Versuchsplanung, die Details der mikroskopischen Bildgebung und die Zielmetriken der Bildanalyse von Anfang an feinsäuberlich aufeinander abstimmen“, rät Chen. Einige für Forschende dafür zentrale Überlegungen hat er vor kurzem mit Dr. Susanne Rafelski und Dr. Matheus Viana vom Allen Institute for Cell Science (USA) im Fachjournal Nature Methods festgehalten hat (siehe unten). Forschende können sie als eine Art Checkliste nutzen, wenn sie sich Gedanken über die Analyse ihrer Bilddaten machen.
-
Grundlagen
-
Sollen die Messungen relativ oder absolut sein?
Welche Messungen oder Variablen werden benötigt?
Soll eine Segmentierung oder eine Vorhersage mit Hilfe von Deep Learning erfolgen oder besteht ein bestimmtes Quantifizierungsziel?
-
-
Grenzen
-
Welche experimentellen Faktoren gibt es zu berücksichtigen?
Welche Mikroskop-Typen und Einstellungen kommen infrage?
Wo liegen die Grenzen der Auflösung?
-
-
Validierung
-
Relative oder absolute Validierung?
Ist das Ziel ein einmaliges oder ein robustes, skalierbares Ergebnis?
Soll die Genauigkeit für das gesamte Bild oder spezifische Merkmale gelten?
Qualitative oder quantitative Fehlerbalken?
-
-
Zeit und Aufwand
-
Was ist der beste Ansatz für die Analyse?
Wieviel Zeit und Aufwand darf die Analyse kosten?
-
Wie gut ist gut genug?
Einen Schlüsselschritt auf dem Weg zur biologisch gültigen Bildanalyse ist die Frage nach der Validierung, also wie genau und präzise die Messungen sein sollen. „Es ist wichtig, dass die Validierung anwendungsgerecht ist und sich an den spezifischen Zielen der Forschung orientiert“, betont Chen. Konkret heißt das, dass Wissenschaftler:innen sich im Voraus überlegen müssen, wie genau ihre Messungen sein sollen und wie viele Fehler akzeptabel sind. Wollen sie etwa Zellteile wie den Zellkern oder die Mitochondrien messen, könnten sie sich die Frage stellen: Ist das exakte Volumen der Organellen relevant, oder geht es vielmehr darum, verschiedene Volumina zu vergleichen? Bei Letzterem spielt das genaue Volumen möglicherweise keine so große Rolle, für den Vergleich müssen die genutzten Computerprogramme aber in verschiedenen Situationen umso besser konstant funktionieren.

Dr. Jianxu Chen
© ISAS / Hannes Woidich
Ein ständiges Anpassen – mithilfe von KI
Doch selbst sehr gute Bilderkennungsalgorithmen arbeiten nicht immer gleich gut, wenn sich beispielsweise die Form der Organellen nur leicht verändert. Die Forschenden müssen ihre Messungen also regelmäßig überprüfen und validieren, einzelne Parameter anpassen oder im schlimmsten Fall sogar ein ganz anderes Programm verwenden. „Bleiben solche Anpassungen unberücksichtigt, kann dies zu grundlegenden Missinterpretationen biologischer Ergebnisse führen“, warnt Chen. Eine Lösung sieht er im Deep Learning, eine Form des maschinellen Lernens, die automatisch Muster und Merkmale aus großen Datenmengen extrahieren kann. Seine Arbeitsgruppe hat bereits ein Plug-in basierend auf Deep Learning für die Segmentierung von Immunzellen (s. Infobox) entwickelt. Bei der Validierung könnte Deep Learning Forschenden zukünftig etwa dabei helfen, Leistungsabfälle in ihren Bildanalysealgorithmen automatisiert zu erkennen.
Segmentierung
In der biomedizinischen Forschung kann es hilfreich sein, verschiedene Strukturen wie Zellen in einem Bild zu erkennen und voneinander abzugrenzen. Diesen Prozess nennt man Segmentierung. Lange war es üblich, die Aufnahmen dafür einzeln durchzugehen und per Hand zu annotieren (zuzuordnen). Mit steigenden Datenmengen und Bildgrößen greifen Forschende heute jedoch zunehmend auf Deep Learning basierte Programme zurück. Diese sind nicht nur deutlich schneller, sondern auch wesentlich objektiver und genauer. Trotzdem dienen die händischen Annotationen häufig noch als Trainingsgrundlage für diese Deep-Learning-Algorithmen. Im Vergleich zu mit biologischer Grundwahrheit trainierten Modellen, kommt es bei solchen, die mit manuellen Annotationen trainiert wurden, zu über 30 Prozent Fehlern (siehe Sonneck 2023). Unter der biologischen Grundwahrheit verstehen die KI-Expert:innen die optimale Annäherung an die objektive Realität, die mit einer einzelnen Analyse nur schwer zu bestimmen ist. Statt mit manuellen Annotationen, ermitteln Expert:innen die biologische Grundwahrheit mithilfe experimentell-computergestützten Analysen.
Die Zukunft der Bildanalyse ist interdisziplinär
Doch Chen und seinem Team geht es nicht nur darum, bessere Wege für die Bildanalyse zu finden. Ihnen liegt viel daran sicherzustellen, dass sich ihre Methoden sowohl in der Biologie als auch in der Computertechnologie verstehen und anwenden lassen. „Wir brauchen mehr interdisziplinäre Zusammenarbeit zwischen Forschenden im Labor- und den KI-Expert:innen, am besten schon früh in der jeweiligen Ausbildung“, fordert Chen. Für diejenigen, die es nicht erwarten können, loszulegen, aber keine Spezialist:innen für Bildverarbeitung in der Nähe haben, empfiehlt Chen Online-Lernmaterialien wie den Bioimaging Guide oder Ressourcen über das Deutsche Netzwerk für Bioinformatik-Infrastruktur - de.NBI. Für spezielle Fragen gibt es auch ein Forum bei der Plattform GitHub. „Nur durch Zusammenarbeit kann sich die quantitative Mikroskopie-Bildanalyse wirklich weiterentwickeln", resümiert Chen.
Das Bundesministerium für Bildung und Forschung (BMBF) fördert die MSCoreSys-assoziierte Nachwuchgruppe AMBIOM – Analysis of Microscopic BIOMedical Images unter dem Förderkennzeichen 161L0272.
Zur Abbildung: Prompt = „I am creating a picture illustration for my article with name “The Art of Balancing: Accuracy in Image Analysis”. It is a press article about how to do bioimage analysis validation. Could you help make an illustration, as normal as possible, and as simple as possible?” Seitens ChatGPT liegen ausschließlich folgende Informationen zur Abbildung vor: „Here is the illustration representing the concept of „The Art of Balancing: Accuracy in Image Analysis” for your article. It features a balance scale with a magnifying glass on one side and a checkmark on the other, symbolizing the equilibrium between analysis and accuracy in bioimage validation.”
(Cheyenne Peters)