Dortmund, 12. August 2025
Wenn Forschende Gewebe untersuchen, um etwa Krankheitsmechanismen auf den Grund zu gehen, müssen sie oft zwischen zwei grundsätzlichen Ansätzen wählen: Entweder sie markieren Substanzen gezielt und untersuchen beispielsweise mit mikroskopischen Verfahren, wie viele davon im Gewebe vorhanden sind. Diese quantitative Analyse setzt allerdings voraus, dass sie bereits wissen, wonach sie suchen. Oder die Wissenschaftler:innen nutzen Methoden wie die bildgebende Massenspektrometrie, um zu sehen, was, beispielsweise an Stoffwechselprodukten, überhaupt im Gewebe vorkommt. Aber aus diesen qualitativen Daten ließ sich bisher nur schlecht die Menge der Stoffe ablesen. Eine Forschergruppe mit ISAS-Beteiligung hat nun eine Methode verfeinert, die sowohl eine qualitative als auch quantitative Untersuchung ermöglicht – und das erstmals gleich für eine ganze Klasse an Substanzen.
Das Team um Prof. Dr. Bernhard Spengler von der Justus-Liebig-Universität Gießen und Prof. Dr. Sven Heiles, Leiter der Arbeitsgruppe Lipidomics am ISAS, kombinierte dazu zwei Analysetechniken: die bildgebende AP-SMALDI MSI (Atmospheric-Pressure Scanning Microprobe Matrix-Assisted Laser Desorption/Ionization Mass Spectrometry Imaging) und die nano-HILIC MS/ MS (nanoflow Hydrophilic Interaction Liquid Chromatography tandem Mass Spectrometry). Sie nutzten diese Kombinationsmethode, um die molekularen Vorgänge bei der Schistosomiasis, einer vernachlässigten Tropenkrankheit mit weltweit über 200 Mio. Erkrankten, zu untersuchen. Ihre Ergebnisse haben die Forschenden in der Fachzeitschrift Analytical Chemistry veröffentlicht.
Molekulare Einblicke in die Schistosomiasis
Schistosomiasis, auch als Bilharziose bekannt, wird ausgelöst, wenn Menschen mit Wasser in Kontakt kommen, in denen Larven von Schistosoma-Würmern leben. Die winzigen Parasiten dringen durch die Haut in den Körper und legen Eier, die sich oft in den Organen ablagern, insbesondere in der Leber. Das menschliche Immunsystem reagiert darauf, indem es Granulome (kugelförmige Gewebestrukturen) um die Eier bildet. Diese sollen die Parasiten eigentlich verkapseln, um sie an der Ausbreitung zu hindern. Die Abwehrreaktion führt jedoch häufig zu Entzündungen, die etwa eine Leberfibrose, eine chronischen Vernarbung des Organs, verursachen können. Was dabei auf molekularer Ebene abläuft, war bisher schwierig zu erfassen.
In ihrer Studie konzentrierten sich die Forschenden auf eine bestimmte Gruppe von Fetten, die Glykosphingolipide (GSLe). Diese Zuckerlipide sind ein wichtiger Bestandteil der Zellmembran. Sie sind wie ein Lollipop gebaut: mit einem fettlöslichen „Stiel“, genannt Ceramid, der in der Membran verankert ist, sowie einem wasserlöslichen Zuckerkopf, der nach außen ragt. GSLe spielen eine wichtige Rolle bei der Kommunikation zwischen Zellen und bei Immunreaktionen. Sie wirken als sogenannte Immunmodulatoren: Antikörper, körpereigene Zellen und Immunzellen erkennen GSLe und reagieren darauf mit einer Immunantwort. Doch bislang ließ sich bildgebend nicht erfassen, wie viele dieser Moleküle in einem spezifischen Gewebe vorhanden sind.
Lesetipp
Luh, D., Heiles, S., Roderfeld, M., Grevelding, C.G., Roeb, E., Spengler, B.
(2024) Hepatic Topology of Glycosphingolipids in Schistosoma mansoni-Infected Hamster. Analytical Chemistry, 96(16):6311-6320.
Endlich eine chemische Abbildung mit relativen Mengenangaben
In ihrer Methode vereinte das Team um Spengler und Heiles die chemische Analyse der nano-HILIC MS/MS mit der hochauflösenden Bildgebung der AP-SMALDI MSI, um das Lebergewebe von gesunden und mit Schistosoma-infizierten Hamstern zu untersuchen. Bei AP-SMALDI-MSI tastet ein feiner Laserstrahl das Gewebe Punkt für Punkt ab und löst Moleküle heraus, die vom Massenspektrometer identifiziert wurden. So entsteht ein chemisches Bild des Gewebes, das der Aufnahme einer Wärmebildkamera ähnelt, aber statt Temperaturen die räumliche Verteilung bestimmter Moleküle zeigt. Durch die Optimierung der Bildpunktauflösung der AP-SMALDI MSI-Methode auf bis zu drei Mikrometer – etwa ein Zwanzigstel der Dicke eines menschlichen Haares – konnten die Wissenschaftler sogar feinste Strukturen innerhalb der Granulome erkennen und damit etwa Antigene und körpereigene GSL bei der Parasiteninfektion auseinanderhalten.
Der entscheidende Fortschritt: Mit ihrer Methode konnten die Forscher nicht nur sehen, wo sich GSL im Gewebe der Tiere befanden, sondern auch ihre relativen Mengen in verschiedenen Geweberegionen vergleichen – ein wichtiger Schritt über die bloße Bildgebung hinaus. Konkret identifizierte das Team 60 verschiedene GSLArten und stellte fest, dass 50 davon in infiziertem Gewebe verstärkt auftraten. 44 dieser Moleküle waren direkt mit Schistosomiasis-bedingter Granulombildung verbunden.
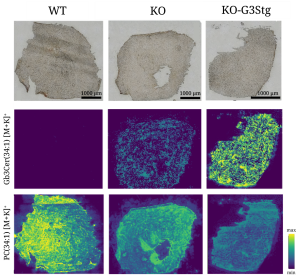
Lipidablagerungen im Herzen bei einem Mausmodell für Morbus Fabry: Die Abbildung zeigt Herzgewebe von Mäusen in drei Gruppen: Links stammt es von gesunden Wildtyp-Tieren (WT). In der Mitte handelt es sich um Knockout- Mäuse (KO), bei denen das Gen für das Enzym α-Galactosidase A gezielt ausgeschaltet wurde. Dieses Enzym baut normalerweise die Glykosphingolipide (GSLe) ab, ist bei Menschen mit Morbus Fabry aber verringert. In der rechten Spalte stammen die Proben von Knockout-Mäusen, die zusätzlich vermehrt GSLe produzieren (KO-G3Stg), wodurch sich die Krankheitsmerkmale verstärken. Die obere Reihe zeigt die Gewebe unter dem Lichtmikroskop. Die mittlere und untere Reihe stellt jeweils dieselben Schnitte mithilfe bildgebender Massenspektrometrie dar: In der mittleren Reihe hebt sich das krankheitsrelevante GSL Gb3Cer 34:1 farblich ab, die untere zeigt zum Vergleich das im Normalzustand vorkommende Zellmembranlipid PC 34:1. Durch den Farbverlauf von minimaler bis zu maximaler Intensität der Messergebnisse deutlich zu erkennen: Je stärker die Morbus-Fabry-typischen Bedingungen ausgeprägt sind, desto mehr schädliche GSLe lagern sich im Herzgewebe ab.
© Sven Heiles
Mehr als nur ein Bild – ein Paradigmenwechsel
Die Arbeit stellt einen Paradigmenwechsel dar: weg von der reinen qualitativen Bildgebung („Was ist wo?“) hin zur quantitativen Analyse („Wie viel ist wo?“). Die entwickelte Methode eröffnet nicht nur neue Einblicke in die Pathologie der Schistosomiasis, sondern hat das Potenzial für verschiedene Bereiche der Biomedizin, in denen die räumliche Verteilung von Lipiden und anderen Biomolekülen von Bedeutung ist: Am ISAS arbeitet Heiles mit seinem Team etwa an Analyseverfahren für den Einsatz bei der genetischen Lipidspeichererkrankung Morbus Fabry. Bei Patient:innen kommt es dabei zu einer langfristig schädlichen Ansammlung von GSLe. Diesen Prozess besser verstehen und analysieren zu können, dürfte perspektivisch den Grundstein für neue Therapiemöglichkeiten legen.
(Ute Eberle)