In diesem Projekt nutzen die Forschenden die Kernspinmagnetresonanzspektroskopie (Nuclear Magnetic Resonance, NMR-Spektroskopie) für die Untersuchung von unbekannten biologischen Prozessen des Stoffwechsels (Metabolismus). Diese Methode eignet sich insbesondere für die Analyse des Metaboloms, also der Gesamtheit der Stoffwechselprodukte eines Organismus oder einer Zelle. Mithilfe der NMR-Spektroskopie lassen sich strukturelle Informationen über Moleküle auf der Nanometerskala bestimmen. In diesem Projekt kommt die NMR-Spektroskopie sowohl für zielgerichtete (targeted) als auch nicht-zielgerichtete (non-targeted) Metabolom-Analysen zum Einsatz. Die Informationen aus gezielten Untersuchungen von festgelegten Metaboliten-Sets können etwa zur Frühdiagnose von Erkrankungen oder zur Überwachung von Arzneimitteltherapien beitragen. Nicht-gerichtete Analysen finden bei der Untersuchung biochemischer Netzwerke und unbekannter Stoffwechselwege Anwendung.
Metabolomische Netzwerkmodelle für unvollständige Reaktionswege
Entlang eines Stoffwechselwegs rufen Enzyme normalerweise sukzessive Transformationen hervor. Unvollständige Spezifität, Instabilität oder Reaktivität mancher Enzyme können allerdings zu einer fehlerhaften chemischen Umsetzung führen. Diese unbeabsichtigten Veränderungen werden in einem gesunden Stoffwechsel durch metabolomische Seitenreaktion kompensiert oder korrigiert. Wenn deren Funktion jedoch gestört ist, besteht die Möglichkeit, dass „nicht-kanonische“ Metabolite entstehen, also untypische Varianten von Metaboliten, die Erkrankungen auslösen können. Derzeit sind metabolomische Seitenreaktionen noch weitestgehend unerforscht und es gibt kaum Möglichkeiten, potenzielle Seitenreaktionen in einem Reaktionsweg gezielt zu lokalisieren. Diese Prozesse zu verstehen sowie mögliche Reparaturenzyme zu identifizieren, könnte daher ein großes Potenzial für die Therapie von Erkrankungen darstellen.
Um potenzielle unvollständige Reaktionswege zu identifizieren, konstruieren die Forschenden am ISAS mithilfe der NMR-Spektroskopie und Massenspektrometrie Netzwerkmodelle spezifischer Metabolite in bestimmten Teilbereichen der Stoffwechselwege. In weiteren Schritten wollen sie mögliche zugrunde liegende enzymatische Reaktionen und Enzyme von Reparaturmechanismen identifizieren.
Optimierung der NMR-Spektroskopie
Die Untersuchungen mittels NMR-Spektroskopie sind nicht-invasiv und benötigen nur wenig Probenvorbereitung. Im Vergleich zur Massenspektrometrie weisen sie allerdings eine geringere Empfindlichkeit auf. Deshalb arbeiten die Wissenschaftler:innen daran, die Analysemethode hinsichtlich der Sensitivität zu optimieren. Gleichzeitig verfolgen sie das Ziel, auch die Empfindlichkeit für Proben mit begrenzter Masse und Volumen zu verbessern.
3D-Modelle der Zellen
Ein weiterer Schwerpunkt des Projekts liegt auf der Entwicklung neuer in-vitro-Methoden. Der biologische 3D-Druck ermöglicht es, gezielt und flexibel Testmodelle für spezifische biologische Fragestellungen zu erzeugen. Solche Modelle kombinieren die Forschenden mit mikrofluidischen Elementen, Lab-on-a-Chips, sowie mit dem Einsatz von Zellmodellen in Form von Spheroiden. So können sie Analysen an Systemen durchzuführen, die zum Beispiel die physiologischen Bedingungen einer Erkrankung simulieren.
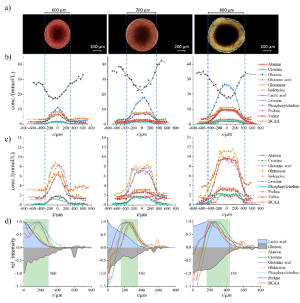
Ergebnisse einer NMR- Spektroskopischen Analyse von 3D- Zellkulturmodellen. (a) zeigt lichtmikroskopische Aufnahmen der Modellzellen. Die Graphen b) und c) bilden die Intensitäten der nachgewiesenen Metabolite ab in den Zellen. d) zeigt ihre relative radiale Verteilung. Hervorgehoben sind Milchsäure (blau), Glukose (grau) sowie der Bereich, in dem die Phosphorylcholin-Konzentration über der Hälfte ihres Maximums liegt.
© ISAS / Roland Hergenröder
Teilen
Ausgewählte Publikationen
Metabolomics, Bd. 21, Nr. 5, 2025
Alwahsh M, Alejel R, Hamadneh L, Aleidi SM, Marchan R, Hasan A, Jasim S, Saqallah FG, Al-Kouz S, Hussein B, Alhusban AA, Al-Hiari Y, Al-Qirim T,…
Identification of potential biomarkers of triton WR-1339 induced hyperlipidemia: NMR-based plasma metabolomics approach and gene expression analysis
https://doi.org/10.1007/s11306-025-02318-z
Analytical Chemistry, Bd. 97, Nr. 4, 2025, S. 1983-1991
Bahti A, Telfah A, Hergenröder R, Suter D.
NMR Spectral Editing, Water Suppression, and Dipolar Decoupling in Low-Field NMR Spectroscopy Using Optimal Control Pulses and Multiple-Pulse Sequence
https://doi.org/10.1021/acs.analchem.3c05226
Journal of Physical Chemistry B, Bd. 128, Nr. 46, 2024, S. 11417-11425
Telfah A, Al Bataineh Q, Al-Essa K, Al-Sawalmih A, Telfah M, Gogiashvili M, Bahti A, Majer G, Hergenröder R.
1H and 13C NMR and FTIR Spectroscopic Analysis of Formic Acid Dissociation Dynamics in Water
https://doi.org/10.1021/acs.jpcb.4c04701
Current Pharmaceutical Analysis, Bd. 20, Nr. 7, 2024, S. 485-499
Alwahsh M, Abumansour H, Althaher AR, Hergenröder R.
Metabolic Profiling Techniques and Their Application in Cancer Research
https://doi.org/10.2174/0115734129317614240809053901
Scientific Reports, Bd. 14, Nr. 1, 2024, S. 14806
Al-Wahsh MI, Nimer RM, Dahabiyeh LA, Hamadneh L, Hasan A, Alejel R, Hergenröder R.
NMR-based metabolomics identification of potential serum biomarkers of disease progression in patients with multiple sclerosis
https://doi.org/10.1038/s41598-024-64490-x
Molecular Biology Reports, Bd. 51, Nr. 1, 2024
Alwahsh M, Al-Doridee A, Jasim S, Awwad O, Hergenroeder R, Hamadneh L.
Cytotoxic and molecular differences of anticancer agents on 2D and 3D cell culture
https://doi.org/10.1007/s11033-024-09669-1
Journal of Cancer, Bd. 15, Nr. 13, 2024, S. 4047-4058
Alwahsh M, Hamadneh Y, Marchan R, Dahabiyeh LA, Alhusban AA, Hasan A, Alrawabdeh J, Hergenröder R, Hamadneh L.
Glutathione and Xanthine Metabolic Changes in Tamoxifen Resistant Breast Cancer Cell Lines are Mediated by Down-Regulation of GSS and XDH and Correlated to Poor Prognosis
https://doi.org/10.7150/jca.96659
Journal of Applied Polymer Science, Bd. 141, Nr. 17, 2024
Ahmad AA, Al-Bataineh QM, Bani-Salameh AA, Al Omari RH, Telfah A.
Self-cleaning antireflected surfaces based on treated PEO/SiO2 nanocomposite films
https://doi.org/10.1002/app.55275
Journal of Applied Polymer Science, Bd. 141, Nr. 29, 2024
Al-Bataineh QM, Ahmad AA, Alakhras LA, Telfah A.
pH-responsivity and photoconductivity for the organic mixed ionic electronic conductor of PANI/PSS composite films
https://doi.org/10.1002/app.55656
Sensors and Actuators A: Physical, Bd. 370, 2024
Al-Bataineh QM, Telfah AD, Tavares CJ, Hergenröder R.
Modeling and analysis of discrete particle detection in wide-field surface plasmon resonance microscopy
https://doi.org/10.1016/j.sna.2024.115266
Materials Chemistry and Physics, Bd. 317, 2024
Al-Bataineh QM, Migdadi AB, Ahmad AA, Brincoveanu O, Mocanu A, Toader G, Telfah AD.
Cobalt-doped SnS2 nanoplates for high-efficiency catalysis applications
https://doi.org/10.1016/j.matchemphys.2024.129184
Journal of Applied Polymer Science, Bd. 141, Nr. 14, 2024
Telfah A, Abu-Zurayk R, Al-Bataineh QM, Tavares CJ, Foadian F, Etzkorn J.
Optical and electrical analysis of polyethylene oxide/disodium hexachloroplatinate complex composite films
https://doi.org/10.1002/app.54980
Applied Surface Science, Bd. 649, 2024
Al-Bataineh QM, Telfah AD, Tavares CJ, Hergenröder R.
Wide-field surface plasmon resonance microscope based on polyethylene oxide/polyacrylic acid brushes
https://doi.org/10.1016/j.apsusc.2023.159189
Journal of Ocular Pharmacology and Therapeutics, Bd. 40, Nr. 1, 2024, S. 78-88
Telfah A, AL-Akhras M, Alshheamat H, Mousa MS, Jum'h I, Albawab A, Tolstik E, Dierks J, Hergenroeder R.
Dissociation Kinetics and Antimicrobial Activity of Ofloxacin Antibiotic in Artificial Tears Via 1H-NMR, Raman, and UV-Vis Spectroscopic Analysis
https://doi.org/10.1089/jop.2023.0019
Journal of Alloys and Compounds, Bd. 971, 2024
Migdadi A, Al-Bataineh QM, Ahmad AA, Al-Khateeb H, Telfah A.
Titanium dioxide/reduced graphene oxide nanocomposites as effective photocatalytic for hazardous 4-nitrophenol
https://doi.org/10.1016/j.jallcom.2023.172794
Applied Physics A: Materials Science and Processing, Bd. 130, Nr. 1, 2024
Telfah A, Al-Bataineh QM, Ahmad AA, Bani‑Salameh AA, Alsaad AM, Sabirianov RF.
Modulated transparent conductive zinc oxide films for efficient water splitting
https://doi.org/10.1007/s00339-023-07176-x
Organic Electronics, Bd. 124, 2024
Telfah A, Al-Bataineh QM, Ahmad AA, Abu-Zurayk R, Tavares CJ, Etzkorn J, Foadian F.
Organic mixed ion-electron conductive composite films based on polyacrylic acid/polyaniline
https://doi.org/10.1016/j.orgel.2023.106933
Scientific Reports, Bd. 13, 2023, S. 1-8
Al-Bataineh QM, Telfah A, Tavares CJ, Hergenröder R.
Surface plasmon coupling between wide-field SPR microscopy and gold nanoparticles
https://doi.org/10.1038/s41598-023-49583-3
Journal of Applied Polymer Science, Bd. 140, Nr. 46, 2023
Al-Bataineh QM, Ahmad AA, Alakhras LA, Telfah A.
Ionic-electronic coupling in PEO-PEDOT/potassium triflate composite films for organic mixed conductivity enhancement
https://doi.org/10.1002/app.54691
Clinical Ophthalmology, Bd. 17, 2023, S. 3719-3728
Al-Dwairi R, Ahmad AA, Aleshawi A, Bani-Salameh AA, Aljarrah IA, Al-Bataineh QM, Al Beiruti S, Alshami AO, Rusen E, Toader G, Hergenröder R.
Silicone Oil Utilized in Pars Plana Vitrectomy for Patients with Advanced Proliferative Diabetic Retinopathy: Physico-Chemical and Optical Properties
https://doi.org/10.2147/OPTH.S447099
Polymer, Bd. 288, 2023, S. 126440
Telfah A, Kalfe-Yildiz A, Bataineh QMA, Jum'h I, Tavares CJ, Hergenröder R.
Thermal-dependent morphological evolution effect on ion transportation in polyethylene oxide films
https://doi.org/10.1016/j.polymer.2023.126440
Scientific Reports, Bd. 13, 2023
Telfah A, Bahti A, Kaufmann K, Ebel E, Hergenröder R, Suter D.
Low-field NMR with multilayer Halbach magnet and NMR selective excitation
https://doi.org/10.1038/s41598-023-47689-2
Journal of Molecular Structure, Bd. 1292, 2023
Telfah M, Al-Akhras M, Telfah A, Jum´h I, Ababneh R, Bustanji Y, Al-Hiari Y, Hergenröder R.
19F- and 1H-NMR investigations of ofloxacin fluoroquinolone tethered with silver nanoparticles as synergistic antibiotic combinations
https://doi.org/10.1016/j.molstruc.2023.136024
Journal of Molecular Structure, Bd. 1292, 2023, S. 136081
Telfah A, Shari'ah NA, Ababneh R, Bahti A, Al-Akhras M, Al-Hiari Y, Jum'h I, Abu-Dahab R, Telfah M, Bataineh QMA, Hergenröder R.
1H-NMR analysis of fluoroquinolone (pyridopyrrole quinoxaline, PPQ) conjugated to gold nanoparticles for synergistic anticancer drug design
https://doi.org/10.1016/j.molstruc.2023.136081
Journal of Materials Science, Bd. 58, Nr. 44, 2023, S. 16942-16953
Khan RAA, Alsaad AM, Zulfqar A, Mateen M, Al Bataineh Q, Al-Anzi BS, Abd-Rabboh HS, Ashraf GA, Telfah A, Luo M.
A simulation study on the effect of polymer–NP interaction strength on the glass transition temperature and phase separation in polymer nanocomposites
https://doi.org/10.1007/s10853-023-09074-2
Journal of Materials Science: Materials in Electronics, Bd. 34, Nr. 32, 2023
Alzoubi F, Ahmad AA, Aljarrah IA, Migdadi A., Al-Bataineh QM.
Localize surface plasmon resonance of silver nanoparticles using Mie theory
https://doi.org/10.1007/s10854-023-11304-x
Journal of Materials Science: Materials in Electronics, Bd. 34, Nr. 20, 2023
Ahmad AA, Alakhras LA, Al-Bataineh QM, Telfah A.
Impact of metal doping on the physical characteristics of anatase titanium dioxide (TiO2) films
https://doi.org/10.1007/s10854-023-10948-z
Journal of Electronic Materials, Bd. 53, Nr. 1, 2023, S. 338-346
Ahmad AA, Aljarrah IA, Al-Bataineh QM, Ababneh R, Telfah A.
Photoluminescence, Optical, and Electrical Properties of Bis(8-Hydroxyquinoline) Zinc and Tris-(8-Hydroxyquinoline) Aluminum Organometallics and Their Films
https://doi.org/10.1007/s11664-023-10788-x
Surfaces and Interfaces, Bd. 39, 2023
Al-Bataineh QM, Ahmad A, Aljarrah I, Telfah A.
Optical and electrical properties of poly(4-styrenesulfonic acid)-polyacrylic acid polyelectrolyte brushes incorporating with potassium permanganate
https://doi.org/10.1016/j.surfin.2023.102923
Sensors, Bd. 23, Nr. 9, 2023, S. 1-11
Al-Bataineh QM, Telfah AD, Shpacovitch V, Tavares CJ, Hergenröder R.
Switchable Polyacrylic Acid Polyelectrolyte Brushes for Surface Plasmon Resonance Applications
https://doi.org/10.3390/s23094283
Biointerface Research in Applied Chemistry, Bd. 13, Nr. 2, 2023
Al-Akhras M, Telfah M, Shakhatreh MN, Telfah A, Mousa MS, Narayanaswamy V, Obaidat IM.
Optical and Chemical Investigations of PEO Thin Films Incorporated with Curcumin Nanoparticle: Effect of Film Thickness
https://doi.org/10.33263/BRIAC132.143
Nanomaterials, Bd. 13, Nr. 7, 2023
Alsaad AM, Al-Bataineh QM, Qattan IA, Aljarrah IA, Bani-Salameh AA, Ahmad AA, Albiss BA, Telfah A, Sabirianov RF.
Physicochemical Properties of Organic Molecular Ferroelectric Diisopropylammonium Chloride Thin Films
https://doi.org/10.3390/nano13071200
Biosensors, Bd. 13, Nr. 4, 2023
Sharar N, Wüstefeld K, Talukder RM, Skolnik J, Kaufmann K, Giebel B, Börger V, Nolte F, Watzl C, Weichert F, Hergenröder R, Shpacovitch V.
The Employment of the Surface Plasmon Resonance (SPR) Microscopy Sensor for the Detection of Individual Extracellular Vesicles and Non-Biological Nanoparticles
https://doi.org/10.3390/bios13040472
Journal of Alloys and Compounds, Bd. 939, 2023
Ribeiro JM, Rodrigues FJ, Correia FC, Pudza I, Kuzmin A, Kalinko A, Welter E, Barradas NP, Alves E, LaGrow AP, Bondarchuk O, Welle A, Telfah A,…
The influence of Sb doping on the local structure and disorder in thermoelectric ZnO:Sb thin films
https://doi.org/10.1016/j.jallcom.2023.168751
Physica B: Condensed Matter, Bd. 654, 2023
Telfah A, Al-Bataineh QM, Salameh B, Ahmad AA, Alsaad AM, Sabirianov RF.
Dielectric relaxation, optical and structural characterizations of complex composite films based on polyethylene oxide doped by low concentration of iodine
https://doi.org/10.1016/j.physb.2023.414649
Nanomaterials, Bd. 13, Nr. 6, 2023, S. 970
Jum'h I, Abu-Safe HH, Ware ME, Qattan IA, Telfah A, Tavares CJ.
Surface Atomic Arrangement of Aluminum Ultra-Thin Layers Grown on Si(111)
https://doi.org/10.3390/nano13060970
Metabolites, Bd. 13, Nr. 3, 2023, S. 352
Migdadi L, Sharar N, Jafar H, Telfah A, Hergenroeder R, Woehler C.
Machine Learning in Automated Monitoring of Metabolic Changes Accompanying the Differentiation of Adipose-Tissue-Derived Human Mesenchymal Stem Cells Employing 1H-1H TOCSY NMR
https://doi.org/10.3390/metabo13030352
Optik, Bd. 273, 2023, S. 170388
Alsaad A, Ahmad AA, Migdadi AB, Telfah A.
Optical, electrical and morphological studies of complex composite films of rubidium bis(2-methyllactato) borate monohydrate doped with iodine
https://doi.org/10.1016/j.ijleo.2022.170388
Journal of Materials Science, Bd. 58, Nr. 6, 2023, S. 2634-2646
Alsaad AM, Migdadi AB, Ahmad AA, Al-Bataineh QM, Telfah A.
Physicochemical characterizations of synthesized polyethylene oxide/boron carbide nanocomposite films
https://doi.org/10.1007/s10853-023-08204-0
Journal of Experimental & Clinical Cancer Research, Bd. 42, Nr. 1, 2023
Keller M, Rohlf K, Glotzbach A, Leonhardt G, Lueke S, Derksen K, Demirci O, Goecener D, AlWahsh M, Lambert J, Lindskog C, Schmidt M, Brenner W,…
Inhibiting the glycerophosphodiesterase EDI3 in ER-HER2+breast cancer cells resistant to HER2-targeted therapy reduces viability and tumour growth
https://doi.org/10.1186/s13046-022-02578-w
EXCLI Journal, Bd. 22, 2023, S. 146-168
Alwahsh M, Farhat J, Talhouni S, Hamadneh L, Hergenroeder R.
Bortezomib advanced mechanisms of action in multiple myeloma, solid and liquid tumors along with its novel therapeutic applications
https://doi.org/10.17179/excli2022-5653
Water, Air, & Soil Pollution , Bd. 234, Nr. 1, 2023
Jum'h I, Abu-Aleqa R, Jumah R, Tavares C, Telfah A.
Efficiency of TiO2/Fe2NiO4 Nanocomposite in Photocatalytic Degradation of Acid Orange 7 (AO7) Under UV Irradiation
https://doi.org/10.1007/s11270-022-05978-y
Materials Chemistry and Physics, Bd. 293, 2023
Al Bataineh Q, Telfah M, Abu-Zurayk R, Benchaabane A, Tavares C, Telfah A.
Nano-SnO2/polyaniline composite films for surface plasmon resonance
https://doi.org/10.1016/j.matchemphys.2022.126816
Jordan Journal of Pharmaceutical Sciences, Bd. 16, Nr. 2, 2023, S. 469
Bahti A, Telfah A, Sharar N, Jafar H, Hergenröder R.
Nuclear Magnetic Resonance for Targeted Metabolomics and Biochemical Sensor
https://doi.org/10.35516/jjps.v16i2.1519
Physica B: Condensed Matter, Bd. 651, 2022, S. 414587
Sadiq D, Al Bataineh Q, Telfah A, Muneer WA, Ahmad AA, Tavares C, Hergenröder R.
Effect of AlNPs distribution on the optical and electrical properties of PANI/AlNPs nanocomposite films
https://doi.org/10.1016/j.physb.2022.414587
Journal of Applied Polymer Science, Bd. 139, Nr. 46, 2022, S. e53138
Al Bataineh Q, Telfah A, Ahmad AA, Bani-Salameh AA, Abu-Zurayk R, Hergenröder R.
E/Z reversible photoisomerization of methyl orange doped polyacrylic acid-based polyelectrolyte brush films
https://doi.org/10.1002/app.53138
Physica B: Condensed Matter, Bd. 646, 2022
Telfah A, Al Bataineh Q, Mousa MS, Ababneh A, Sadiq D, Tavares CJ, Hergenröder R.
HR MAS NMR, dielectric impedance and XRD characterization of polyethylene oxide films for structural phase transitions
https://doi.org/10.1016/j.physb.2022.414353
Journal of Ocular Pharmacology and Therapeutics, Bd. 38, Nr. 10, 2022, S. 717-727
Al-Dwairi R, Ahmad AA, Aleshawi A, Bani-Salameh A, Aljarrah I, Al-Bataineh Q, Mohidat H, Al Sharie AH.
Optical and Physical Properties of Silicone Oil Extracted from the Vitreous of Patients Who Underwent Vitrectomy for Retinal Detachment
https://doi.org/10.1089/jop.2022.0081
Biosensors, Bd. 12, Nr. 12, 2022
Al Bataineh Q, Shpacovitch V, Sadiq D, Telfah A, Hergenröder R.
Surface Plasmon Resonance Sensitivity Enhancement Based on Protonated Polyaniline Films Doped by Aluminum Nitrate
https://doi.org/10.3390/bios12121122
Journal of Materials Science: Materials in Electronics, Bd. 33, Nr. 35, 2022, S. 26225-26235
Al Bataineh Q, Bani-Hani WT, Ahmad AA, Alsaad A, Telfah A.
Promising electrochemical catalytic steel electrodes structure coated by ZnO films for water treatment and water-splitting applications
https://doi.org/10.1007/s10854-022-09307-1
Physica B: Condensed Matter, Bd. 645, 2022, S. 414224
Al Bataineh Q, Ahmad AA, Alsaad A, A.B. Migdadi AB, Telfah A.
Correlation of electrical, thermal, and crystal parameters of complex composite films based on polyethylene oxide (PEO) doped by copper sulfate (CuSO4)
https://doi.org/10.1016/j.physb.2022.414224
Journal of Magnetism and Magnetic Materials, Bd. 562, 2022, S. 169822
Telfah A, Ghellab T, Baaziz H, Charifi Z, Alsaad A, Sabirianov R.
First-principles calculations to investigate strong half-metallic ferromagnetic and thermoelectric sensibility of LiCrX (X= S, Se, and Te) alloys
https://doi.org/10.1016/j.jmmm.2022.169822
Nature Communications, Bd. 13, Nr. 1, 2022, S. S68
Jeanclos E, Schlötzer J, Hadamek K, Yuan-Chen N, Al-Wahsh MI, Knitsch R, Fratz S, Yesiyurt-Gerhards D, Frankenbach T, Engelmann D, Keller A, Kaestner…
Glycolytic flux control by drugging phosphoglycolate phosphatase
https://doi.org/10.1038/s41467-022-34228-2
Applied Physics A - Materials Science & Processing, Bd. 128, Nr. 11, 2022, S. 965-973
Al Bataineh Q, Ahmad AA, Aljarrah I, Alsaad A, Telfah A.
Hidden impurities in transparent conducting oxides: study of vacancies-related defects and impurities in (Cu–Ni) co-doped ZnO films
https://doi.org/ttps://doi.org/10.1007/s00339-022-06028-4
Journal of Electronic Materials, Bd. 51, Nr. 11, 2022, S. 6540–6546
Ahmad AA, Al Bataineh Q, Bani-Salameh AA, Alsaad A, Telfah A.
Physicochemical Properties of Sodium Bis(2-methyllactato)borate Films Doped with Iodine for Photonic Applications
https://doi.org/10.1007/s11664-022-09895-y
Materials Chemistry and Physics, Bd. 290, 2022, S. 126387
Al Bataineh Q, A.B. Migdadi AB, Telfah A, Ahmad AA, Alsaad A, Tavares C.
Physical and chemical characterization of polyaniline (PANI)/indium tin oxide nanoparticles (ITONPs) nanocomposite films
https://doi.org/10.1016/j.matchemphys.2022.126387
Polymer Bulletin, Bd. 372, Nr. 9, 2022, S. 9611-9625
Telfah A, Al Bataineh Q, Tolstik E, Ahmad AA, Mohammad Alsaad A, Ababneh R, Tavares C, Hergenröder R.
Optical, electrical and chemical properties of PEO: I2 complex composite films
https://doi.org/10.1007/s00289-022-04508-4
Surfaces and Interfaces, Bd. 33, 2022, S. 102202
Ahmad AA, Al Bataineh Q, Bani-Salameh AA, Telfah A.
Optical, optoelectronic, structural, and chemical characterizations of (PEO-PVA)/MWCNT nanocomposite films
https://doi.org/10.1016/j.surfin.2022.102202
Polymers for Advanced Technologies, Bd. 34, Nr. 1, 2022, S. 110-119
Ahmad MJA, Telfah A, Al Bataineh Q, Tavares C, Hergenröder R.
Nanoparticles positioning effect on properties of (PS-PANI/NiNPs) nanocomposite films
https://doi.org/10.1002/pat.5870
Journal of Materials Science: Materials in Electronics, Bd. 33, Nr. 26, 2022, S. 20778–20789
Al Bataineh Q, Ahmad AA, Alsaad A, Aljarrah I, Telfah A.
Structural, chemical, morphological and optical properties of thin films based on potassium bis(2-methyllactato) borate hemihydrate doped by iodine
https://doi.org/10.1007/s10854-022-08887-2
Polymer Bulletin, Bd. 80, 2022, S. 7533-7543
Aljarrah I, Bani-Salameh AA, Ahmad AA, Al Bataineh Q, Alsaad A, Al-Akhras M, Telfah A.
Effect of UV-illumination on refractive index of PMMA/metal oxide nanocomposite films
https://doi.org/10.1007/s00289-022-04409-6
Anti-Cancer Agents in Medicinal Chemistry, Bd. 22, Nr. 19, 2022, S. 3304-3321
Hallaq T, Al-Hiari Y, Kasabri V, AlBashiti R, Alalawi S, Telfah A.
In vitro Antiproliferative Properties of Lipophililic-Acid Chelating Fluoroquinolones and TriazoloFluoroquinolones with 7-dihaloanilinosubstitution
https://doi.org/10.2174/1871520622666220513154744
Journal of Materials Science: Materials in Electronics, Bd. 33, Nr. 22, 2022, S. 17434–17445
Al Bataineh Q, Ababneh R, Bahti A, Bani-Salameh AA, Tavares C, Telfah A.
Effect of hydrogen-related shallow donor on the physical and chemical properties of Ag-doped ZnO nanostructures
https://doi.org/10.1007/s10854-022-08513-1
Applied Physics A - Materials Science & Processing, Bd. 128, Nr. 8, 2022
Alsaad A, Aljarrah I, Ahmad AA, Al Bataineh Q, Shariah A, Al-Akhras M, Telfah A.
The structural, optical, thermal, and electrical properties of synthesized PEO/GO thin films
https://doi.org/10.1007/s00339-022-05829-x
Journal of Applied Polymer Science, Bd. 139, Nr. 26, 2022, S. 1-11
Jum'h I, Telfah A, Mousa MS, Ahmad MJA, Tavares C, Hergenröder R.
XPS, UV–Vis, XRD, and PL spectroscopies for studying nickel nanoparticle positioning effect on nanocomposite film properties
https://doi.org/10.1002/app.52433
Journal of Applied Polymer Science, Bd. 139, Nr. 25, 2022
Al Bataineh Q, Telfah A, Ahlmann N, Tolstik E, Tavares CJ, Hergenröder R.
Photoisomerization kinetics of a novel photoswitchable film based on methyl red doped with sodium hexachloroplatinate hosted in polyethylene oxide
https://doi.org/10.1002/app.52387
Photochemistry and Photobiology , Bd. 98, Nr. 4, 2021, S. 831-842
Ahmad AA, Al Bataineh Q, Al-Nawafleh DM, Telfah A.
Photoisomerization Kinetics of Photoswitchable Thin Films Based on Nanostructure/Molecular Layers of AlN-AO7
https://doi.org/10.1111/php.13535
Photochemistry and Photobiology , Bd. 98, Nr. 4, 2021, S. 823-830
Ahmad AA, Al Bataineh Q, Al-Nawafleh DM, Al-Nawafleh AM, Telfah A.
Synthesis and Characterization of Thin Films Based on Azobenzene Derivative Anchored to CeO2 Nanoparticle for Photoswitching Applications
https://doi.org/10.1111/php.13534
The European Physical Journal Plus, Bd. 137, 2022
Ahmad AA, Alsaad A, Aljarrah I, Al Bataineh Q, Telfah A.
Optical, electronic, and structural properties of different nanostructured ZnO morphologies
https://doi.org/10.1140/epjp/s13360-022-02967-2
Polymer Bulletin, 2022, S. 5433-5446
A.B. Migdadi AB, Ahmad AA, Mohammad Alsaad A, Al Bataineh Q, Telfah A.
Electrical and thermal characterizations of synthesized composite films based on polyethylene oxide (PEO) doped by aluminium chloride (AlCl3)
https://doi.org/10.1007/s00289-022-04329-5
Computational and Structural Biotechnology Journal, Bd. 20, 2022, S. 2965-2977
Migdadi LYH, Telfah A, Hergenröder R, Wöhler C.
Novelty Detection for Metabolic Dynamics Established On Breast Cancer Tissue Using 2D NMR TOCSY Spectra
https://doi.org/10.1016/j.csbj.2022.05.050
Physica B: Condensed Matter, Bd. 634, 2022
Al-Akhras M, Shakhatreh MN, Chamroukhia H, Telfah A, Tavares C.
Optical, electrical and morphological properties of (PANI/CSA-PEO)/(AgNPs-AgNO3) nanocomposite films
https://doi.org/10.1016/j.physb.2021.413636
Asian Pacific Journal of Cancer Prevention, Bd. 23, Nr. 5, 2022, S. 1529-1537
Qashou E, Al-Hiari Y, Kasabri V, AlBashiti R, Alalawi S, Telfah A, AlHadid A.
Antiproliferative Activities of Lipophililic Fluoroquinolones-Based Scaffold Against a Panel of Solid and Liquid Cancer Cell Lines
https://doi.org/DOI:10.31557/APJCP.2022.23.5.1529
Journal of Materials Science: Materials in Electronics, Bd. 33, 2022, S. 14051–14062
Al Bataineh Q, Aljarrah I, Ahmad AA, Alsaad A, Telfah A.
Investigation of the doping mechanism and electron transition bands of PEO/KMnO4 complex composite films
https://doi.org/10.1007/s10854-022-08336-0
Journal of Materials Science: Materials in Electronics, Bd. 33, Nr. 13, 2022, S. 10585–10595
Ahmad AA, Aljarrah I, Telfah M, Alsaad A, Telfah A.
Optical, chemical, electrical, and morphological properties of PEO–Nb-doped KMnO4 thin films
https://doi.org/10.1007/s10854-022-08044-9
Solid State Communications, Bd. 347, 2022
Ghellab T, Charifi Z, Baaziz H, Telfah A, Ababneh R, Alsaad A, Sabirianov R.
Physical properties of LiXH4 (X= B, Al) hydrogen storage materials: ab-initio study
https://doi.org/10.1016/j.ssc.2022.114731
Cancers, Bd. 14, Nr. 2196, 2022
Farhat J, Alzyoud L, Al-Wahsh MI, Al-Omari B.
Structure–Activity Relationship of Benzofuran Derivatives with Potential Anticancer Activity
http://10.3390/cancers14092196
Materials Science in Semiconductor Processing, Bd. 141, 2022, S. 106415
Ghellab T, Baaziz H, Charifi Z, Telfah M, Alsaad A, Telfah A, Hergenröder R, Sabirianov R.
The structural, electronic, optical, thermodynamical and thermoelectric properties of the Bi2Al4Se8 compound for solar photovoltaic semiconductors
https://doi.org/10.1016/j.mssp.2021.106415
Materials Chemistry and Physics, Bd. 280, 2022, S. 125793
Ahmad AA, Al‑Bataineh QM, Aljarrah I, Telfah A.
Electrochemical degradation of methyl red in zinc hydroxide and zinc oxide thin films, physical and chemical activation
https://doi.org/10.1016/j.matchemphys.2022.125793
Journal of Magnetism and Magnetic Materials, Bd. 546, 2021, S. 168919
Ghellab T, Baaziz H, Charifi Z, Latelli H, Ahmad MJA, Telfah M, Alsaad A, Telfah A, Hergenröder R, Sabirianov R.
First-principles calculations of the high-pressure behavior, electronic, magnetic, and elastic properties of praseodymium pnictides: PrX (X = P, As and Bi)
https://doi.org/10.1016/j.jmmm.2021.168919
Cancers, Bd. 14, Nr. 6, 2022
Alwahsh M, Knitsch R, Marchan R, Lambert J, Hoerner C, Zhang X, Schalke B, Lee D, Bulut E, Graeter T, Ott G, Kurz KS, Preissler G, Schölch S, Farhat…
Metabolic Profiling of Thymic Epithelial Tumors Hints to a Strong Warburg Effect, Glutaminolysis and Precarious Redox Homeostasis as Potential Therapeutic Targets
https://doi.org/10.3390/cancers14061564
Journal of Materials Science: Materials in Electronics, Bd. 33, Nr. 8, 2022, S. 5087–5104
A.B. Migdadi AB, Ahmad AA, Alsaad A, Telfah A.
Synthesis, optoelectronic and thermal characterization of PMMA-MWCNTs nanocomposite thin films incorporated by ZrO2 NPs
https://doi.org/10.1007/s10854-022-07699-8
Applied Surface Science, Bd. 574, Nr. 151647, 2021
T. Figueiredo W, Prakash R, G. Vieira C, S. Lima D, Carvalho VE, Soares EA, Buchner S, Raschke H, W. Perez-Lopez O, L.Baptista D, Hergenröder R,…
New insights on the electronic factor of the SMSI effect in Pd/TiO2 nanoparticles
https://doi.org/10.1016/j.apsusc.2021.151647
Polymer Bulletin, Bd. 80, 2022, S. 809-828
Ahmad AA, Bani-Salameh AA, Al Bataineh Q, Jum'h I, Telfah A.
Optical, structural and morphological properties of synthesized PANI-CSA-PEO-based GaN nanocomposite films for optoelectronic applications
https://doi.org/10.1007/s00289-021-04033-w
Heliyon, Bd. 8, Nr. 1, 2022, S. e08683
Ahmad AA, A.B. Migdadi AB, Mohammad Alsaad A, Qattan I, Al Bataineh Q, Telfah A.
Computational and experimental characterizations of annealed Cu2ZnSnS4 thin films
https://doi.org/10.1016/j.heliyon.2021.e08683
Ceramics International, Bd. 48, Nr. 1, 2022, S. 266–277
Dhahri I, Ellouze M, Labidi S, Al Bataineh Q, Etzkorn J, Guermazi H, Telfah A, Tavares CJ, Hergenröder R, Appel T.
Optical and structural properties of ZnO NPs and ZnO–Bi2O3 nanocomposites
https://doi.org/10.1016/j.ceramint.2021.09.101
Polymer Bulletin, Bd. 79, 2021, S. 9881-9905
Ahmad AA, Khazaleh MH, Mohammad Alsaad A, Al Bataineh Q, Telfah A.
Characterization of As-prepared PVA-PEO/ZnO-Al2O3-NPs hybrid nanocomposite thin films
https://doi.org/10.1007/s00289-021-03969-3
Colloids and Surfaces A: Physicochemical and Engineering Aspects, Bd. 628, 2021
Al-Gharram M, Jum'h I, Telfah A, Al-Hussein M.
Highly crystalline conductive electrodeposited films of PANI-CSA/CoFe2O4 nanocomposites
https://doi.org/10.1016/j.colsurfa.2021.127342
Breast Cancer Research , Bd. 28, Nr. 6, 2021, S. 1358-1366
Hamadneh L, Bahader M, Abuarqoub R, Al-Wahsh MI, Alhusban A, Hikamat S.
PI3K/AKT and MAPK1 Molecular Changes Preceding Matrix Metallopeptidases Overexpression during Tamoxifen Resistance Development are Correlated to Poor Prognosis in Breast Cancer Patients
https://doi.org/10.1007/s12282-021-01277-2
Analytical Chemistry, Bd. 93, Nr. 40, 2021, S. 13485–13494
Knitsch R, Al-Wahsh MI, Lambert J, Raschke H, Hergenröder R.
In vitro spatio-temporal NMR metabolomics of living 3D cell models
https://doi.org/10.1021/acs.analchem.1c02221
Computational and Structural Biotechnology Journal, Bd. 19, 2021, S. 5047-5058
Migdadi LYH, Lambert J, Telfah A, Hergenröder R, Wöhler C.
Automated Metabolic Assignment: Semi-Supervised Learning in Metabolic Analysis Employing Two Dimensional Nuclear Magnetic Resonance (NMR)
https://doi.org/10.1016/j.csbj.2021.08.048
Physica Status Solidi (b), Bd. 258, Nr. 10, 2021, S. 2100039
Telfah A, Essaoud SS, Baaziz H, Charifi Z, Alsaad A, Ahmad MJA, Hergenröder R, Sabirianov R.
Density Functional Theory Investigation of Physical Properties of KCrZ (Z = S, Se, Te) Half-Heusler Alloys
https://doi.org/10.1002/pssb.202100039
Optik, Bd. 241, 2021, S. 167014
Alsaad A, Al‑Bataineh QM, Bani-Salameh AA, Ahmad AA, Albiss B, Telfah A, Sabirianov R.
Synthesis and structural, crystallographic, electronic, chemical and optical characterizations of alpha-diisopropylammonium bromide (α-DIPAB) thin films
https://doi.org/10.1016/j.ijleo.2021.167014
Cells, Bd. 10, Nr. 7, 2021, S. 1657
Farhat J, Pandey I, AlWahsh M.
Transcending toward Advanced 3D-Cell Culture Modalities: A Review about an Emerging Paradigm in Translational Oncology
https://doi.org/10.3390/cells10071657
Journal of Magnetic Resonance, Bd. 328, 2021
Bahti A, Telfah A, Lambert J, Hergenröder R, Suter D.
Optimal control pulses for subspectral editing in low field NMR
https://doi.org/10.1016/j.jmr.2021.106993
International Journal of Energy Research, Bd. 45, Nr. 11, 2021, S. 16383-16399
Telfah A, Guendouz D, Charifi Z, Baaziz H, Mohammad Alsaad A, Hergenröder R, Sabirianov R.
Theoretical investigations of alkaline earth hydrides XH2 (X=Ca, Sr, and Ba) for hydrogen storage applications
https://doi.org/10.1002/er.6887
Polymer Bulletin, Bd. 79, 2021, S. 3759-3778
Telfah A, Al-Akhras M, Al-Izzy KA, Ahmad AA, Ababneh R, Ahmad MJA, Tavares C, Hergenröder R.
Dielectric relaxation, XPS and structural studies of polyethylene oxide/iodine complex composite films
https://doi.org/10.1007/s00289-021-03593-1
Green Chemistry, Bd. 23, Nr. 9, 2021, S. 3439-3458
K Verma S, Kumar Panda P, Kumari P, Patel P, Arunima A, Jha E, Husain S, Prakash R, Hergenröder R, Kumar Mishra Y, Ahuja R, S Verma R, Suar M.
Determining factors for the nano-biocompatibility of cobalt oxide nanoparticles: proximal discrepancy in intrinsic atomic interactions at differential vicinage
https://doi.org/10.1039/D1GC00571E
Polymers, Bd. 13, Nr. 5, 2021
Al-Bataineh QM, Ahmad AA, Alsaad A, Aljarrah I, Qattan I, Telfah A.
Effect of Iodine Filler on Photoisomerization Kinetics of Photo-Switchable Thin Films Based on PEO-BDK-MR
https://doi.org/10.3390/polym13050841
Jordan Journal of Biological Sciences , Bd. 14, Nr. 1, 2021, S. 41-49
Zouaghi N, Bensiradj NEH, Cavaleiro C, Nadjemi B, Telfah A.
Antimicrobial Activities of Natural Volatiles Organic Compounds Extracted from Dittrichia viscosa (L.) by Hydrodistillation
https://doi.org/10.54319/jjbs/140107
Heliyon, Bd. 7, Nr. 1, 2021, S. e05952
Al-Bataineh QM, Ahmad AA, Mohammad Alsaad A, Telfah A.
Optical characterizations of PMMA/metal oxide nanoparticles thin films: bandgap engineering using a novel derived model
https://doi.org/10.1016/j.heliyon.2021.e05952